
Alle iLive-Inhalte werden medizinisch überprüft oder auf ihre Richtigkeit überprüft.
Wir haben strenge Beschaffungsrichtlinien und verlinken nur zu seriösen Medienseiten, akademischen Forschungseinrichtungen und, wenn möglich, medizinisch begutachteten Studien. Beachten Sie, dass die Zahlen in Klammern ([1], [2] usw.) anklickbare Links zu diesen Studien sind.
Wenn Sie der Meinung sind, dass einer unserer Inhalte ungenau, veraltet oder auf andere Weise bedenklich ist, wählen Sie ihn aus und drücken Sie Strg + Eingabe.
Die größte 3D-Rekonstruktion eines Fragments des menschlichen Gehirns wurde erstellt
Zuletzt überprüft: 02.07.2025
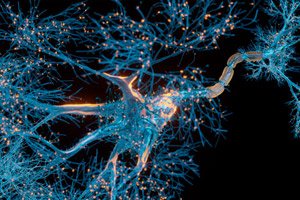 ">
">Ein Kubikmillimeter Hirngewebe mag nicht viel erscheinen. Doch wenn man bedenkt, dass dieses kleine Quadrat 57.000 Zellen, 230 Millimeter Blutgefäße und 150 Millionen Synapsen enthält – insgesamt 1.400 Terabyte an Daten –, haben die Harvard- und Google-Forscher enorme Fortschritte gemacht.
Ein Harvard-Team unter der Leitung von Jeff Lichtman, dem Jeremy R. Knowles-Lehrstuhl für Molekular- und Zellbiologie und neu ernannten Dekan der Fakultät für Naturwissenschaften, und Google-Forscher haben die bislang größte 3D-Rekonstruktion des menschlichen Gehirns auf synaptischer Ebene erstellt. Sie zeigt in lebhaften Details jede Zelle und ihr Netzwerk neuronaler Verbindungen in einem Teil des menschlichen Temporallappens von der Größe eines halben Reiskorns.
Die in der Fachzeitschrift Science veröffentlichte Leistung ist die jüngste in einer fast zehnjährigen Zusammenarbeit mit Wissenschaftlern von Google Research, die Lichtman-Elektronenmikroskopie mit KI-Algorithmen kombiniert, um die hochkomplexe neuronale Verdrahtung von Säugetieren farblich zu kodieren und zu rekonstruieren. Die drei Co-Erstautoren der Arbeit sind der ehemalige Harvard-Postdoc Alexander Shapson-Ko, Michal Januszewski von Google Research und der Harvard-Postdoc Daniel Berger.
Das ultimative Ziel der Zusammenarbeit, die von der BRAIN-Initiative der National Institutes of Health unterstützt wird, besteht darin, eine hochauflösende Karte der neuronalen Konnektivität im gesamten Mäusegehirn zu erstellen. Dazu wären etwa 1.000-mal mehr Daten erforderlich, als sie aus einem Kubikmillimeter menschlicher Hirnrinde gewonnen haben.
Das Wort „Fragment“ ist ironisch gemeint. Ein Terabyte ist für die meisten Menschen eine riesige Menge, aber ein Fragment eines menschlichen Gehirns – nur ein winziger Teil eines menschlichen Gehirns – ist immer noch Tausende von Terabyte groß.“
Jeff Lichtman, Jeremy R. Knowles Professor für Molekular- und Zellbiologie
Die neueste Karte in Science enthält bisher unbekannte Details der Gehirnstruktur, darunter ein spärliches, aber leistungsfähiges Axonnetzwerk, das durch bis zu 50 Synapsen verbunden ist. Das Team stellte außerdem einige Besonderheiten im Gewebe fest, beispielsweise eine geringe Anzahl von Axonen, die ausgedehnte Spiralen bilden. Da die Probe einem Epilepsiepatienten entnommen wurde, ist unklar, ob solche ungewöhnlichen Formationen pathologisch oder einfach nur selten sind.
Lichtmans Forschungsgebiet ist die „Konnektomik“. Ähnlich der Genomik zielt sie darauf ab, vollständige Kataloge der Gehirnstruktur bis hin zu einzelnen Zellen und Verbindungen zu erstellen. Solche vollständigen Karten werden den Weg zu neuen Erkenntnissen über Gehirnfunktionen und -krankheiten ebnen, über die die Wissenschaft noch immer sehr wenig weiß.
Die hochmodernen KI-Algorithmen von Google können Hirngewebe dreidimensional rekonstruieren und kartieren. Das Team hat außerdem eine Reihe öffentlich zugänglicher Tools entwickelt, mit denen Forscher das Konnektom untersuchen und kommentieren können.
„Angesichts der enormen Investitionen, die in dieses Projekt geflossen sind, war es wichtig, die Ergebnisse so zu präsentieren, dass nun auch jemand anderes davon profitieren kann“, sagte Viren Jain, Fellow bei Google Research.
Als nächstes wird sich das Team auf eine Region des Hippocampus der Maus konzentrieren, die für die Neurowissenschaft aufgrund ihrer Rolle beim Gedächtnis und bei neurologischen Erkrankungen von Bedeutung ist.
